菌群多样性组成谱分析
高通量测序分析环境微生物(微生物菌群分析、微生物多样性分析、微生物群落分析)
资费:请电话商谈 联系电话:010-65945597/65919547
手机:13717768726
研究对象:
样品来源:土壤、海洋、活性污泥、动物肠道、粪便、酒曲、窖泥、发酵液、菌剂、食品等;
样品形式:环境样品、基因组DNA、PCR产物;
菌群多样性组成谱测序研究
菌群多样性组成谱测序以细菌/古菌16S rRNA基因可变区、真菌18S rRNA基因可变区或真菌ITS(Internal transcribed spacer)内转录间隔区等微生物特征序列(Signature sequence)为靶点,通过检测序列的变异和数量,反映菌群中各类微生物物种的身份和丰度,从而快速解码菌群物种分布特征,阐明样本间多样性和组成差异,进而发现差异相关物种。
产品特点
1. 直接对菌群样本中的微生物特征序列进行扩增检测,简单快速且性价比高,并克服了绝大部分微生物无法纯培养的难题;
2. 自动化、专业的DNA提取流程,配合高保真DNA聚合酶进行PCR扩增,并严格把控扩增循环数,确保菌群组成客观真实;
3. 使用Illumina MiSeq的小型化测序平台,周期更短,读长更长(2´300 bp),每个样本都能一次性获得数万条序列,即使低丰度物种也能精确定量;
4. 多种物种注释数据库可选,根据样本来源按需选择Greengenes/Silva/HOMD/RDP/Unite/MaarjAM等数据库,更优化物种的分类鉴定;
5. 可对菌群进行代谢功能预测,通过组成数据,解读潜在功能,并能指导后续宏基因组测序研究。
实验流程
微生物组总DNA提取→目标片段PCR扩增→扩增产物检测定量→测序文库制备→上机进行高通量测序
分析流程
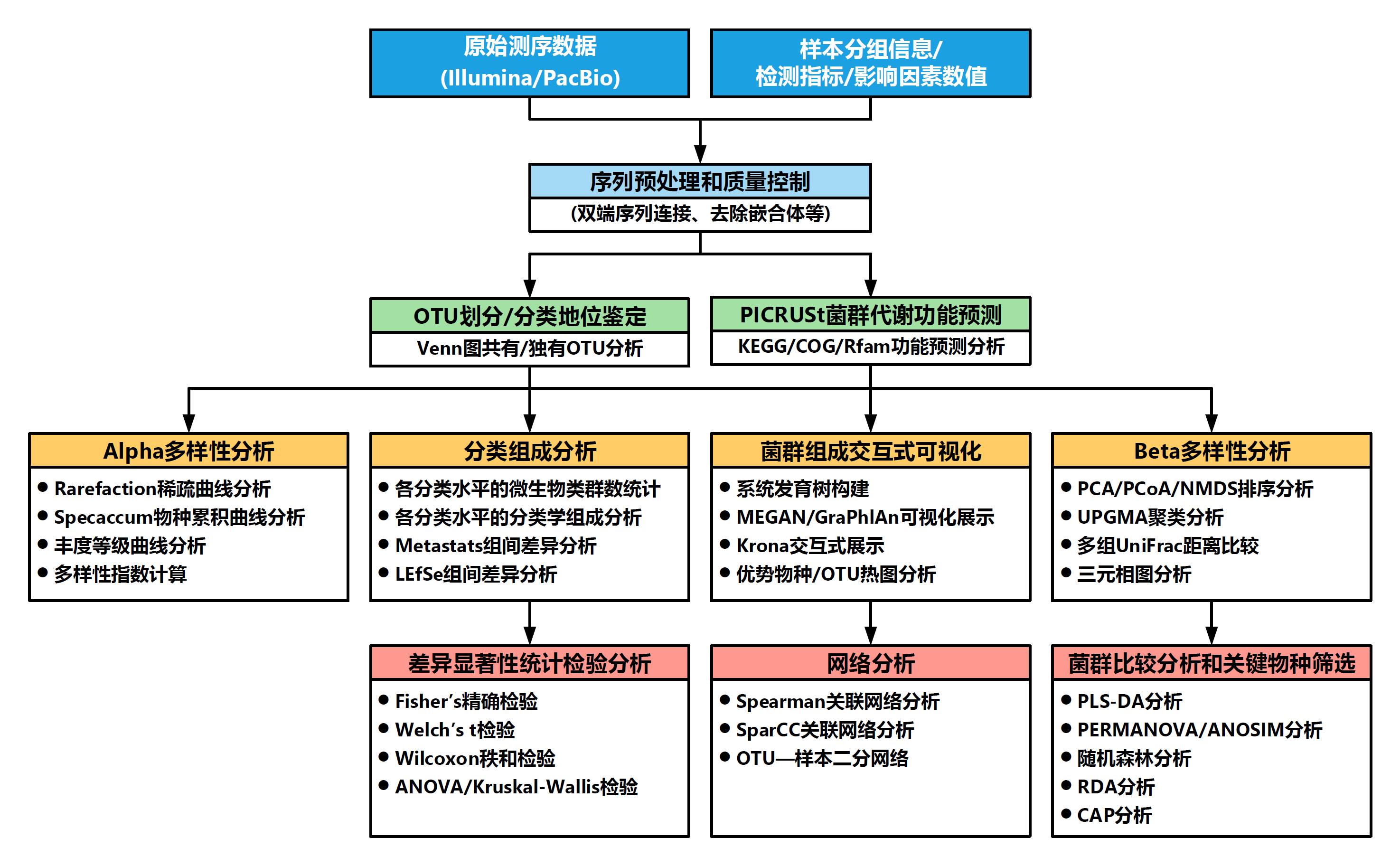
典型分析结果展示
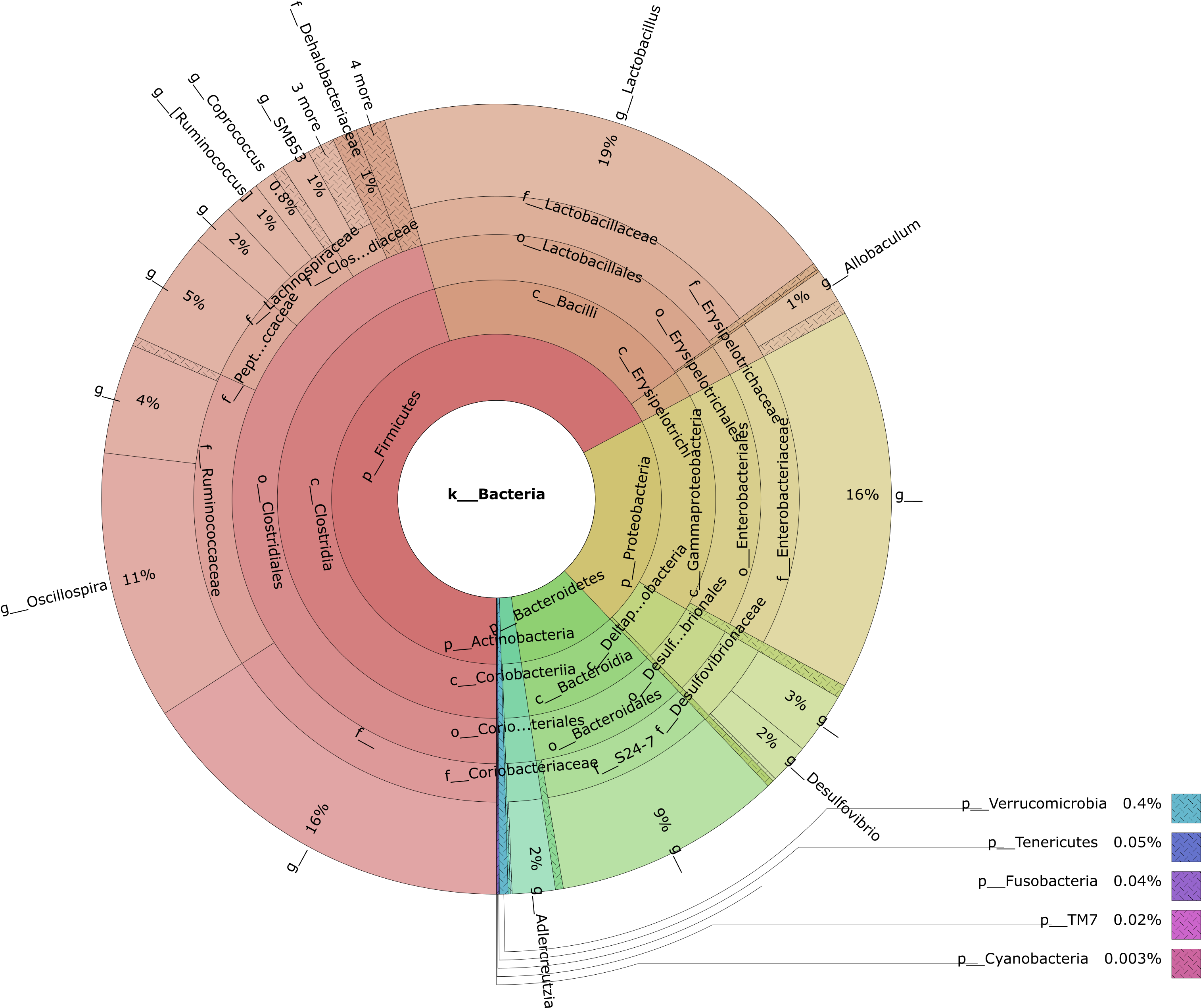
菌群物种分类组成的Krona交互展示图
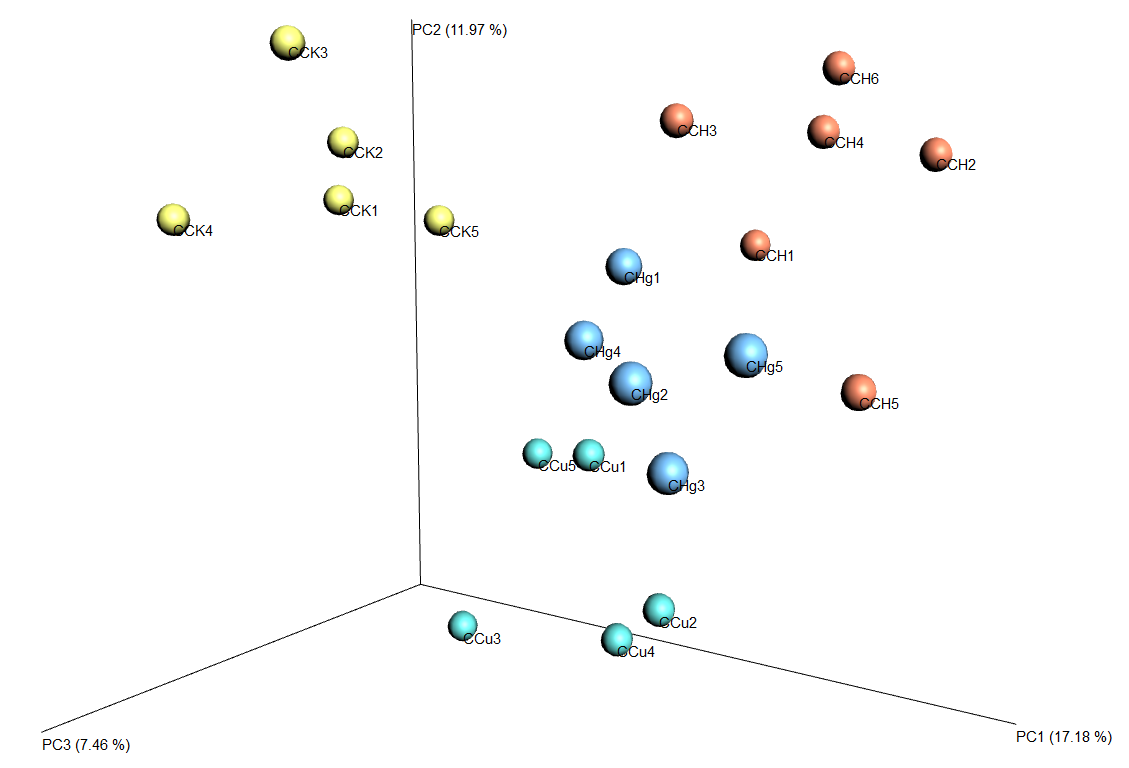
UniFrac PCoA分析的样本三维排序图
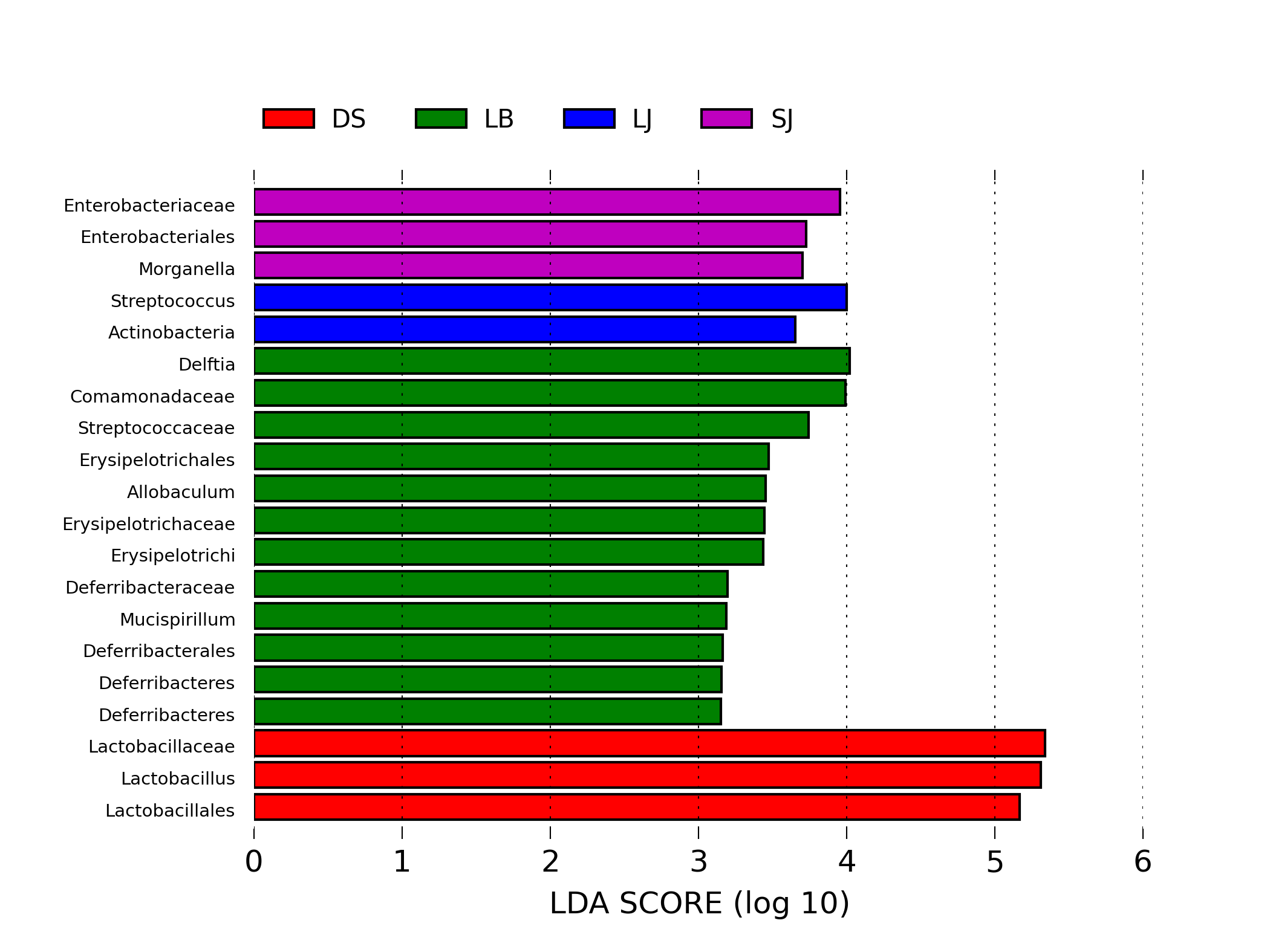
LEfSe组间差异物种的显著性排序图
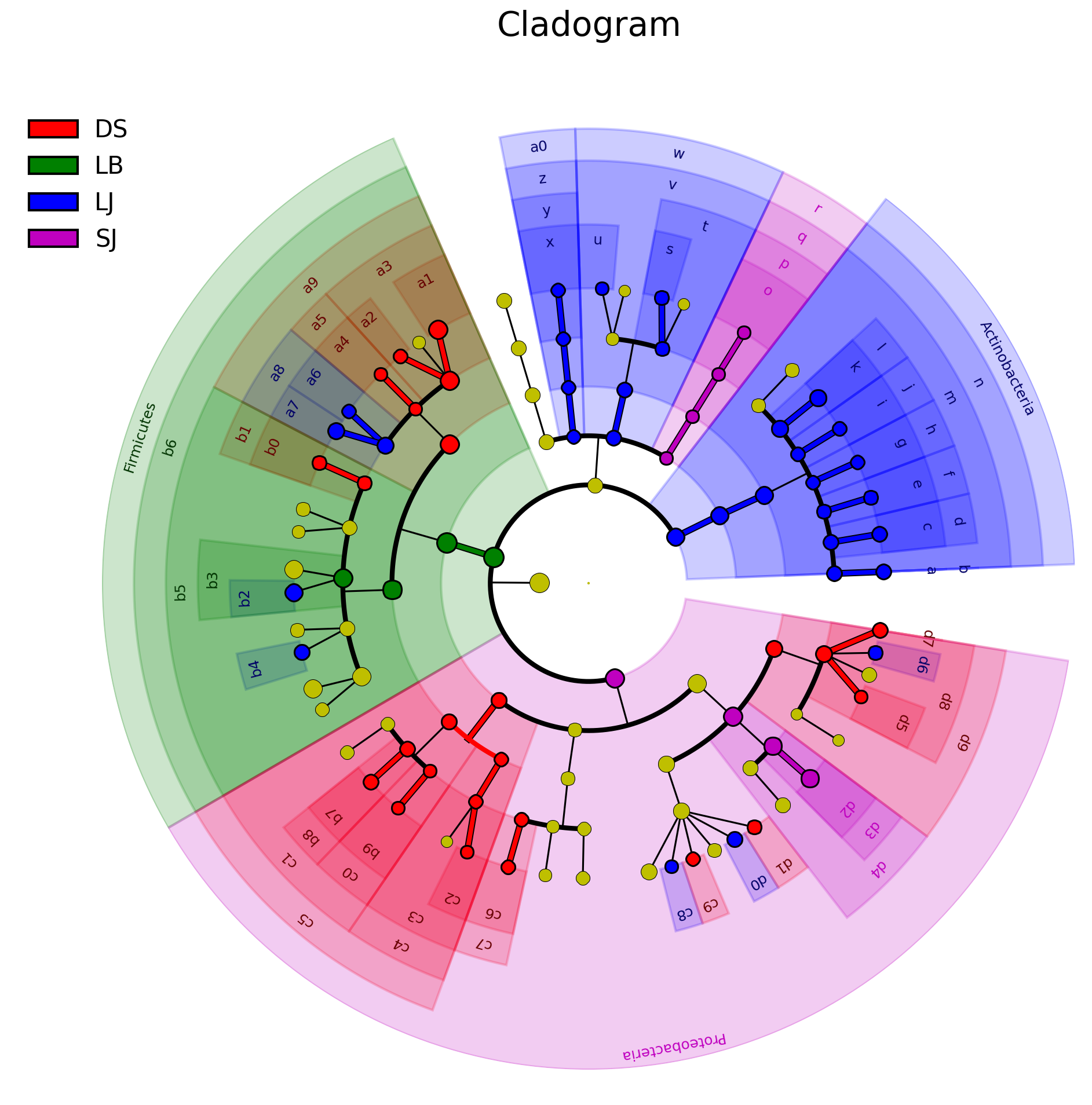
LEfSe组间差异物种的分类等级树图
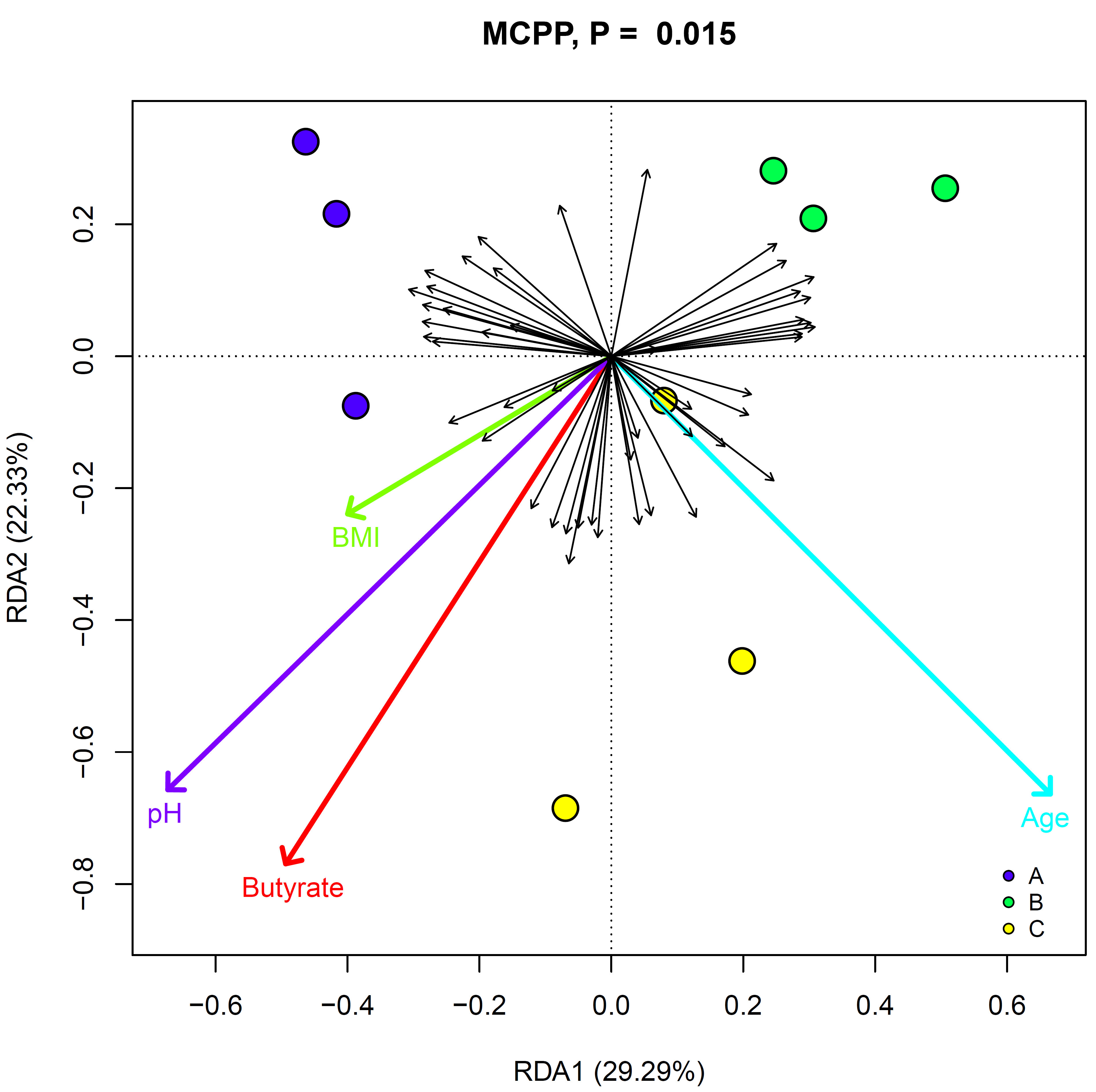
RDA约束排序图
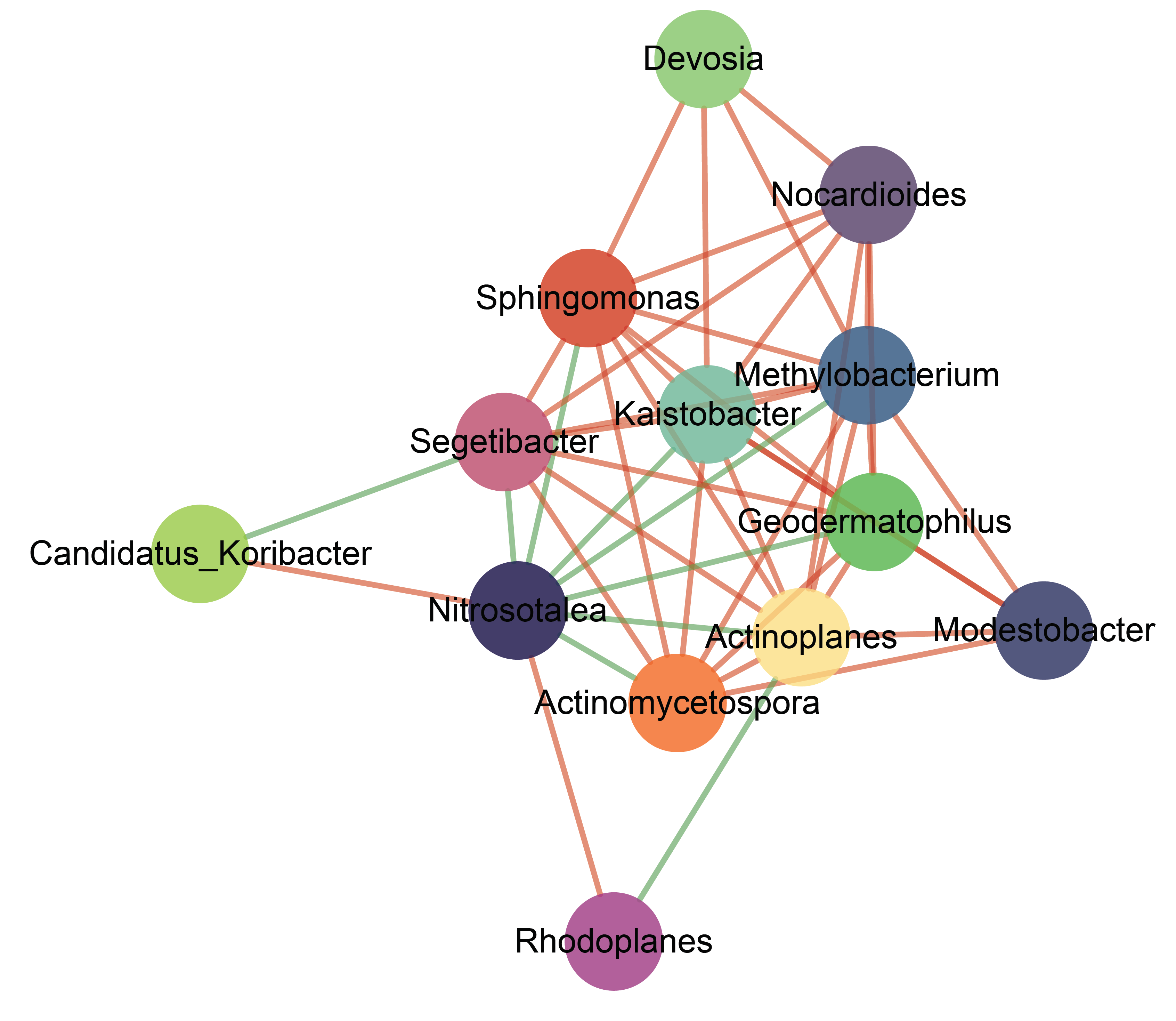
物种互作关联网络图
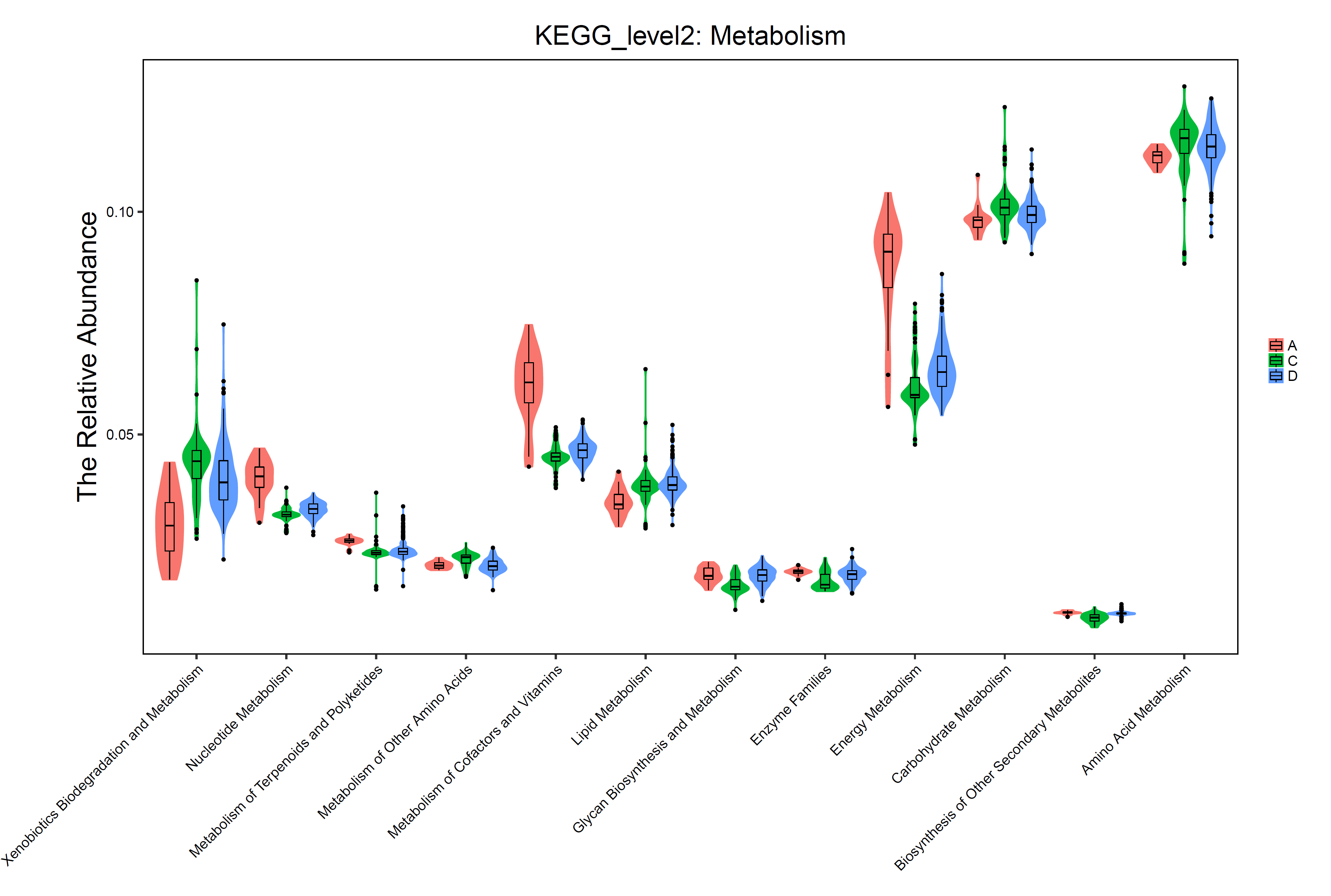
基于16S rRNA基因的PICRUSt功能预测小提琴图
值得一试的深度分析内容
|
分析项目 |
分析要求 |
|
CAP主坐标典型相关分析 |
分组≥ 2 |
|
菌群分型(Enterotype)分析 |
样本≥ 20 |
|
ROC曲线建模验证分析 |
分组≥ 2 |
|
OTU—样本二分网络分析 |
样本≥ 3 |
|
物种——功能一致性Circos分析 |
样本≥ 3 |
|
… |
… |
分析内容
|
分析项目 |
分析 |
备注 |
|
|
基础分析项目 |
|
|
原始测序数据的质控 |
|
|
|
|
1 |
原始测序数据的处理 |
√ |
|
|
|
2 |
疑问序列的剔除 |
√ |
|
|
|
3 |
高质量序列长度分布统计 |
√ |
|
|
|
序列归并和OTU划分 |
|
|
1 |
OTU划分 |
√ |
|
|
|
2 |
OTU分类地位鉴定 |
√ |
|
|
|
3 |
OTU精简和分类鉴定结果统计 |
√ |
|
|
|
4 |
多样本共有OTU的Venn图分析 |
√ |
样本(组)≤ 5 |
|
|
|
|
|
常规分析项目 |
|
|
Alpha多样性分析 |
|
|
1 |
Rarefaction稀疏曲线 |
√ |
|
|
|
2 |
Specaccum物种累积曲线 |
√ |
样本≥ 10 |
|
|
3 |
丰度等级曲线 |
√ |
|
|
|
4 |
Alpha多样性指数计算 |
√ |
|
|
|
分类组成分析 |
|
|
1 |
各分类水平的微生物类群数统计 |
√ |
|
|
|
2 |
各分类水平的分类学组成分析 |
√ |
|
|
|
3 |
Metastats分析 |
√ |
样本(组)≥ 2 |
|
|
4 |
LEfSe分析 |
√ |
分组≥ 2 |
|
|
群落组成交互式可视化 |
|
|
1 |
系统发育树构建 |
√ |
|
|
|
2 |
MEGAN可视化展示 |
√ |
|
|
|
3 |
GraPhlAn可视化展示 |
√ |
|
|
|
4 |
Krona交互式展示 |
√ |
|
|
|
5 |
热图分析 |
√ |
样本≥ 3 |
|
|
Beta多样性分析 |
|
|
1 |
PCA分析 |
√ |
样本≥ 3 |
|
|
2 |
UniFrac-PCoA分析 |
√ |
样本≥ 3 |
|
|
3 |
UniFrac-MDS分析 |
√ |
样本≥ 5 |
|
|
4 |
UniFrac-UPGMA聚类分析 |
√ |
样本≥ 3 |
|
|
5 |
基于UniFrac距离的多组比较和箱线图展示 |
√ |
分组≥ 2 |
|
| |
高级分析项目 |
| |
菌群比较分析和关键物种筛选 |
| |
1 |
RDA分析 |
√ |
样本≥ 3:提供影响因素数值或分组≥ 2 |
| |
2 |
PLS-DA分析 |
√ |
分组≥ 2 |
| |
3 |
Adonis/PERMANOVA分析 |
√ |
样本≥ 3:提供影响因素数值或分组≥ 2 |
| |
4 |
ANOSIM分析 |
√ |
分组≥ 2 |
| |
5 |
随机森林分析 |
√ |
分组≥ 2 |
| |
优势物种互作关联网络分析 |
√ |
样本(组)≥ 3 |
| |
群落代谢功能预测 |
√ |
仅限16S rRNA基因数据 |
送样要求 1
1. 样品类型: DNA
2. 样品需求量:≥500ng
3. 样品浓度: ≥10ng/μL
4. 样品纯度:OD260/280=1.8-2.0并确保DNA无降解
5. 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输。
送样要求 2
1. 样品类型: PCR产物
2. 样品需求量:≥100ng
3. 样品浓度: ≥5ng/μL
4. 样品纯度:OD260/280=1.8-2.0并确保PCR产物无降解
5. 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输。 |